R語言學習-富集分析泡泡圖剛一出品,Y叔就說有硬傷。Y叔是著名富集分析軟件clusterprofiler的原創,而且軟件內集成dotplot,enrichmap,cnetmap (后續也實現這兩個的一步出圖)等畫圖方法,具體看這個教程http://guangchuangyu.github.io/2016/01/go-analysis-using-clusterprofiler/或Biobabble公眾號。
這個意見得重視,不過大夏天的,鍋還是不能背著,回應下Y叔的回應。
一個command出圖,小白上點心可以的
圖是ggplot2畫的(散點圖),在腳本功能描述里有寫。只需要在終端輸入腳本的名字sp_enrichmentPlot.sh
(腳本需在環境變量中)就可以看到腳本功能描述、輸入文件樣例、參數列表和使用例子。
目前腳本的輸入是不支持Windows的Excel格式的,只支持TAB分割的文本文件。之前一直在Linux下工作,文件名都不寫后綴的。后來跟Windows用戶打交道多,為了方便他們打開,強行加了xls后綴,這一點有誤導。
腳本依賴R,這是應該做一個判斷和提示的。但有R卻提示ggplot2包或其它包不存在,是可以用-i TRUE來自動安裝包的。(最近在跟德春合作,完善包的自動檢測和安裝,最后一起整合在分發包。)
Y叔的這兩點確實戳中了當前文檔信息不完善的弱點,也希望大家多反映使用過程中的問題,幫助我們改進文檔。
不過話說回來,如果仔細看了當前腳本的功能描述和參數提示,上點心得小白不只可以一步畫圖,還可以隨意調整樣式。
不只clusterprofiler的用戶需要
如果用clusterprofiler做富集分析,write.table輸出結果,那么輸入文件、R、ggplot2都有了,一步出圖沒有問題。(Windows下沒bash?https://mp.weixin.qq.com/s/g1twNEsPWZb_tZFDyoTqVQ,看這里)
Y叔說既然有了clusterprofiler,一步步在R里面做不是很好,為啥要把數據導出來?
首先,轉換數據、存儲數據這一步是獨立于作圖的。圖只能顯示部分數據(這點Y叔也有提到,用了simplify會好些,但也還會有不少通路),所有富集條目導出作為文章附表,以顯示信息的全面和真實。
其次,即便可以畫出所有富集數據(用一副大圖),也會先對結果做下篩選,一些特別基礎的、極父層的生物富集通路也會選擇不展示,優先展示樣品屬性更相關的。畢竟是看在哪些通路里面富集,不是看在哪些通路里面最富集。所以需要導出數據,做下篩選,然后一步繪圖。
再次,不少人拿到的是已經做好的富集分析結果(可能是clusterprofiler的,也可能是其它軟件的),總不能再導入clusterprofiler繪圖吧,可以有更簡單的方式的。
最后,一個圖做出來,需要反復修改。今天用clusterprofiler做了富集分析,運用dotplot配合不同參數出了圖;過幾天心情一變,想換個風格了,怎么辦?再運行一次clusterprofiler還是加載之前存儲的.Rdata。好像都不太方便,還是用導出的文本一步出圖吧。
關于硬傷
示例圖中沒有overlap不是繪圖腳本的問題,是數據篩選的問題。腳本如實反映篩選出的數據。如果選擇的通路在一個基因集合富集,另外一個不富集,就不顯示;如果選擇的通路是在多個基因集中都富集的,但富集程度不同,是可以顯示的;而且這些通路會優先顯示在圖的上部,下面我會給出例子。
一步出圖也可以定制
說到互動,一步出圖不只可以,而且還可以記錄互動(給每個輸出文件加個唯一的字符串做為文件名的一部分就可以了,不過這個沒用到,之前就沒寫這個參數)。
拿Y叔的數據做個例子
library(clusterProfiler)
library(dplyr)
data(gcSample)
x <- compareCluster(gcSample,'enrichDO')
#獲取數據,取出每個組最富集的10個條目,存儲起來
#不麻煩的吧
x <- x@compareClusterResult
y = x %>% group_by(Cluster) %>%top_n(-10, pvalue)
y = x[x$Description %in% y$Description,]
在樣品名中包含差異基因的數目
#這個不解釋了,前面繪圖教程中提到過
y$Sample <- paste0(y$Cluster,"(",unlist(lapply(strsplit(y$GeneRatio, '/'), function(x)x[2])),")")
#這次存成tsv,省得誤會了
write.table(y,file="enrichment.tsv", sep="\t", col.names=T, row.names=F,quote=F)
首先出第一個圖,很簡單,一個命令,設置下顏色。
+更正下,我這里作弊了一下,之前點的大小都是Gene Count這樣的純數字列來顯示的,不支持GeneRatio這樣的分數形式的列(上一版本,如果GeneRatio出現在橫軸是支持的),做了下修改,也支持了。如果想用之前的代碼重復,把-s GeneRatio改為-s Count。
#具體參數解釋看上一篇公眾號文章
# -C:指定顏色,默認是紅綠,可以是任何兩個R支持的顏色名字
sp_enrichmentPlot.sh -f enrichment.tsv -oSample -T string -v Description -c p.adjust -s GeneRatio -C"'red','blue'" -w 16
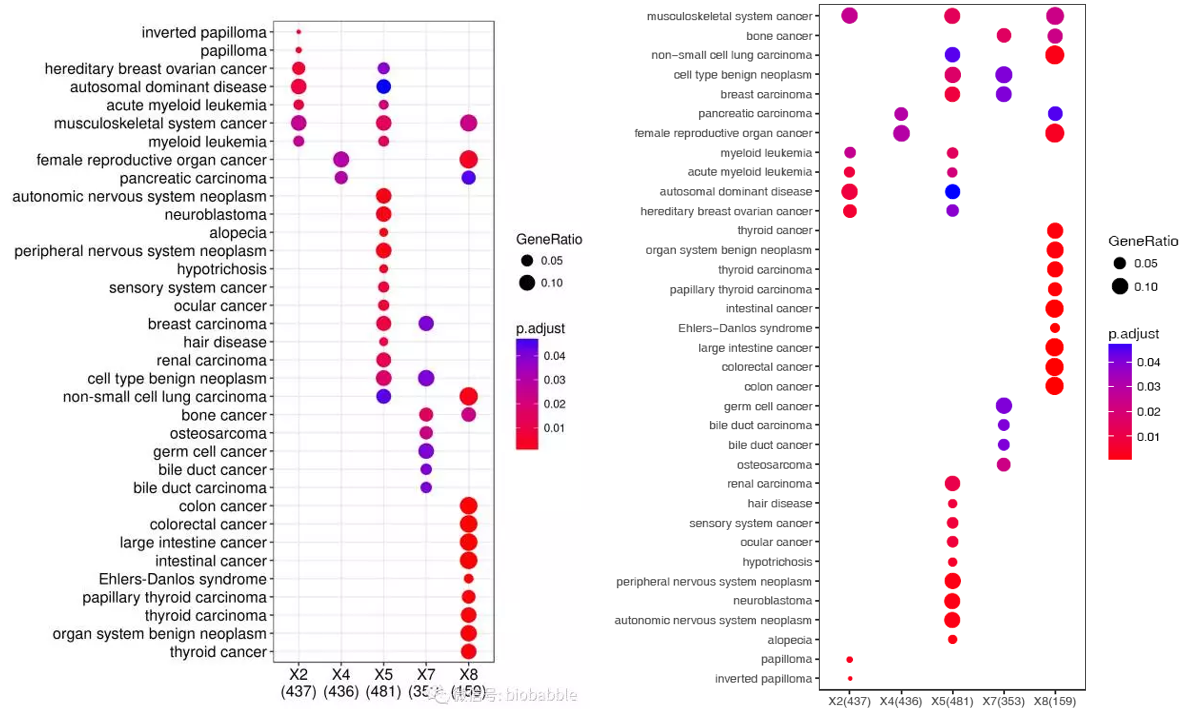
圖略有不同,是因為排序的方式不同。sp_enrichmentPlot.sh設置的是先按照出現在不同樣品最多的條目優先的策略,可以清晰的看到哪些是不同基因集共同富集的,哪些是不同基因集特異富集的。其它的都一樣。當然沒有設置字體參數,字體略小了些。
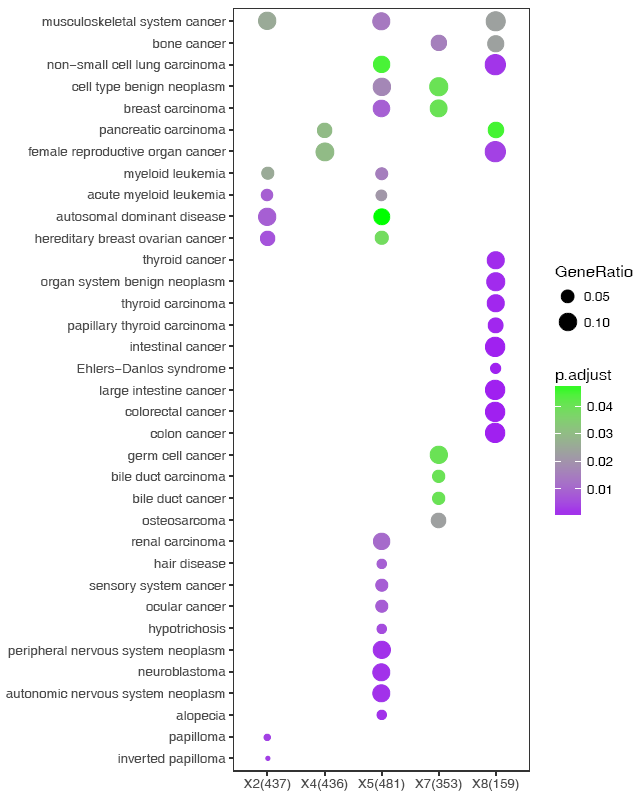
說還想要個紫綠的,也可以,只需改兩個單詞,而不是加一段代碼scale_color_continuous(low='purple',
high='green')。
sp_enrichmentPlot.sh -f enrichment.tsv -oSample -T string -v Description -c p.adjust -s GeneRatio -C"'purple','green'" -w 16
改變下形狀,也可以啊。程序中都留有一個參數可以寫入ggplot2語句,就是下面的-z。那么,ggplot2能修改的樣式也都可以。常用的修改會做成一個參數,不常用的就只能直接寫命令了。能在Rstudio里面敲,那么也能在命令行敲。
#-z:后跟任何合法的ggplot2語句
sp_enrichmentPlot.sh -f enrichment.tsv -oSample -T string -v Description -c p.adjust -s GeneRatio -C"'purple','green'" -w 16 -z "+aes(shape=GeneRatio>0.1)"
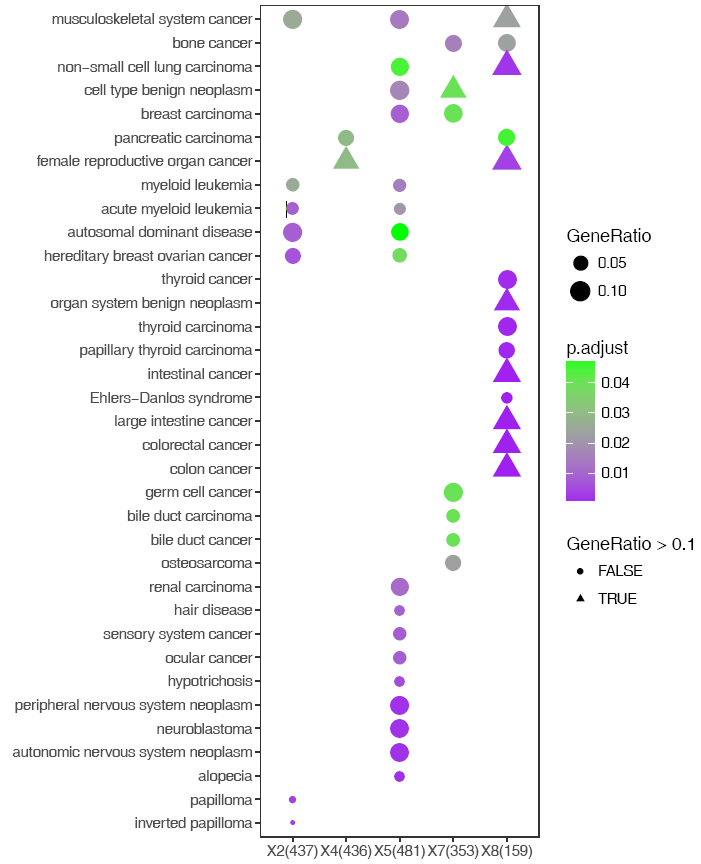
-z “+ any legal ggplot2command”
當然這一步也可以不通過增加ggplot2語句來實現,在數據中加一列就好了,通過-S指定參數。
不加ggplot2語句,只修改命令行參數,sp_enrichmentPlot.sh也可以調整圖例的位置、輸出文件的格式、是否對p.adj取log、是否分面等。
一步作圖的優勢
一步作圖相比于直接寫R代碼有什么好處呢?
1.模塊化好。也就是Y叔提到的數據處理和可視化分開;一步作圖,只是作圖,不做無關的處理,更隨意。
2.易用性強。敲代碼,總不如給改數來得快。
3.重用性強。假如我要做十幾個分開的基因集合呢?一段段復制代碼?改錯了或忘記改某個地方了怎么辦?
4.快速出圖。先快速出個原型,再接著調整。